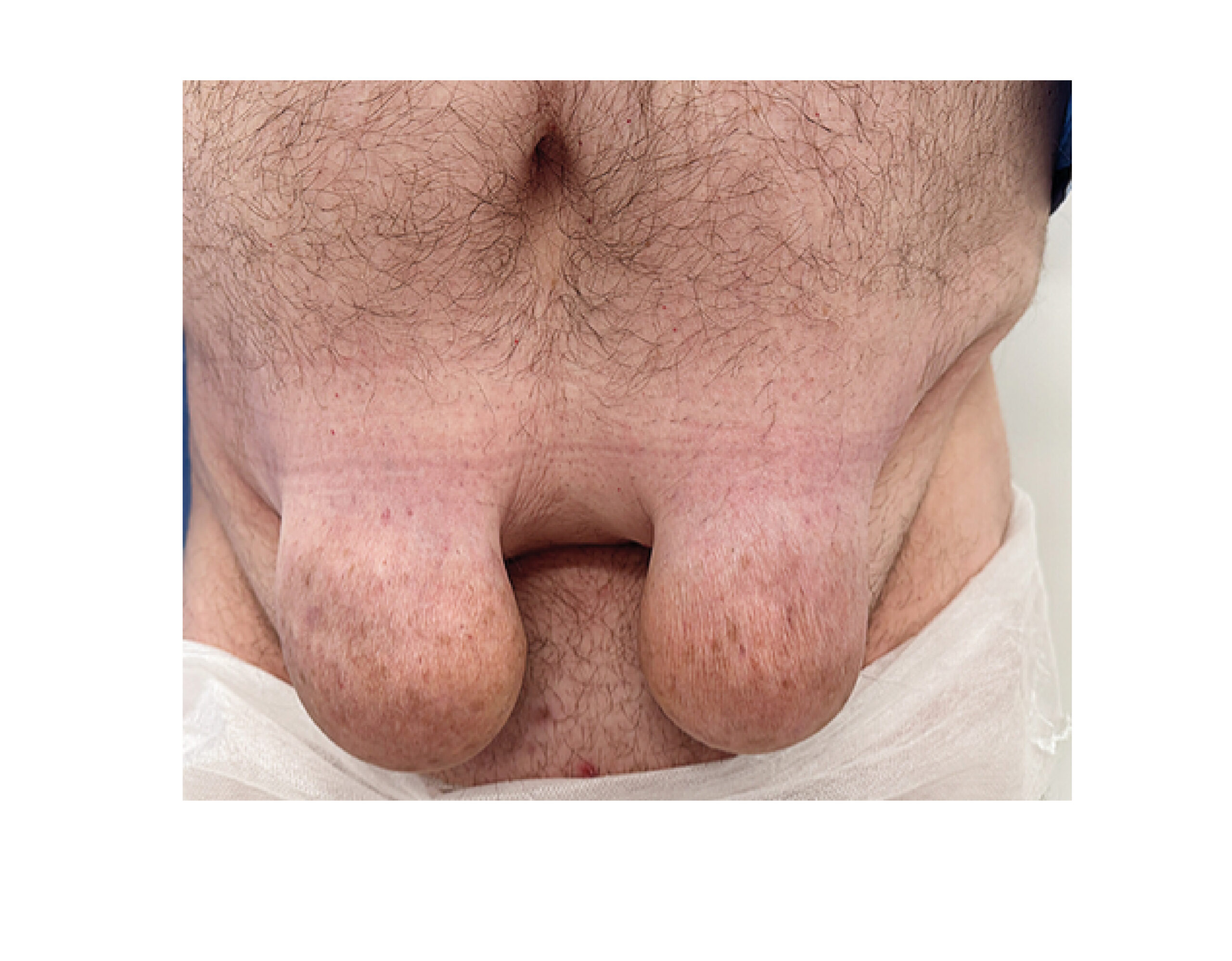
Une équipe de chercheurs lillois vient de développer une nouvelle méthode de séquençage du génome humain qui optimise en termes de temps et de coûts la détermination des mutations causales de maladies génétiques. Testée avec succès dans l’obésité extrême de l’enfant associée à une déficience intellectuelle, cette nouvelle méthodologie appelée CoDE-seq permet en une seule étape le séquençage des mutations ponctuelles de tous les gènes, et la détection très précise des délétions et duplications chromosomiques (appelées CNV). Ces résultats ont été publiés dans Molecular Metabolism.
Le diagnostic génétique souffre d’un grand retard en France. En cause, le manque d’équipes hospitalières en génétique cliniques entraînées à analyser correctement et rapidement le génome, la réglementation, mais aussi la complexité et la lenteur actuelles du processus « remboursé » d’exploration génétique des patients avec une suspicion de maladie génétique, qui associe de manière séquentielle des méthodes obsolètes (ex : le caryotype), à des méthodes incomplètes (ex : puces à ADN) et au séquençage ciblé de l’ADN (en général de quelques gènes seulement). Ce “parcours du combattant” est particulièrement lent (des mois à des années), extrêmement onéreux (des milliers d’euros) et ne bénéficie complètement qu’à une minorité de patients. En 2018 en France, seule une minorité de patients bénéficie d’une exploration complète.
L’équipe lilloise, en collaboration avec la société suisse Roche Diagnostics, et avec les médecins et chercheurs de l’université catholique de Lille, a voulu améliorer le parcours de soins des patients atteints de maladies génétiques en développant une méthode d’analyse du génome plus efficace, moins chère et surtout plus rapide. CoDE-seq (pour Copy number variation Detection and Exome sequencing) combine le criblage de haute qualité de l’ADN des régions codantes des 20 000 gènes humain (exome) au séquençage de centaines de milliers de sondes de 120 nucléotides recouvrant de manière régulière l’ensemble du génome humain permettant l’analyse précise des variants du nombre de copies de gènes (CNV). En effet, si dans la plupart des cas on reçoit deux copies de chaque gène, l’un du père l’autre de la mère (hors chromosomes sexuels), il existe de nombreuses régions chromosomiques ou parfois il manque de l’ADN (délétion) ou il y a de l’ADN surnuméraire (duplication). Dans les deux cas, ces anomalies du nombre de copies de gènes (ou de parties de gènes) peuvent être responsables de maladies génétiques, comme le sont les mutations qui perturbent la fonction des protéines produites par les gènes mutés.
L’équipe lilloise est partie du kit commercial de Roche Diagnostics appelé MedExome qui n’explore que les exons des gènes humains, pour l’enrichir de sondes couvrant tous les chromosomes, en collaboration avec l’équipe américaine de R & D de la société NimbleGen Roche basée à Hamilton. Elle a étudié 82 patients avec une suspicion de forme monogénique d’obésité et/ou de déficience intellectuelle, dont 40 avaient été testés par la méthode de puce à ADN pour la détection des CNV. Non seulement CoDE-seq a pu détecter les 97 CNV identifiés par puce, mais en a identifiés 84 de plus. Sur ces 82 patients, un diagnostic moléculaire a pu être mis en évidence chez 27 d’entre eux (avec l’identification d’une ou deux altérations génétiques pouvant être responsable de la maladie). La moitié de ces diagnostics sont dus à des CNV l’autre moitié à des mutations ponctuelles de l’exome.
Cette nouvelle analyse de l’exome augmenté est rapide (trois semaines, extraction de l’ADN et analyse bio-informatique comprises), et peu onéreuse (< 500€ à ce jour). Elle ouvre des perspectives prometteuses pour le diagnostic moléculaire des maladies génétiques rares.
© Amélie Bonnefond & Philippe Froguel
Source : http://www.cnrs.fr/insb/recherche/parutions/articles2018/p-froguel2.html